ChIP-seq原理
一文读懂 ChIP-seq:从实验原理到结果图分析
1. 什么是 ChIP-seq,它能做什么?
首先,我们来拆解一下这个名字:
- ChIP = Chromatin Immunoprecipitation (染色质免疫共沉淀)
- seq = sequencing (二代测序)
合在一起,ChIP-seq 的核心思想就是:用一个“钩子”钓出与特定蛋白质结合的 DNA 片段,然后通过测序技术,告诉我们这些 DNA 片段在基因组的什么位置。
简单来说,ChIP-seq 就是用来回答“在细胞内,某个蛋白质(比如转录因子、组蛋白)都结合在哪些 DNA 区域?”这个问题的。
目前,ChIP-seq 主要有两大应用方向:
方向一:研究转录因子 (Transcription Factor, TF)
转录因子就像是基因的“开关”。它们是一些能结合到特定基因上游区域的蛋白质,通过这种结合来开启或关闭基因的表达。
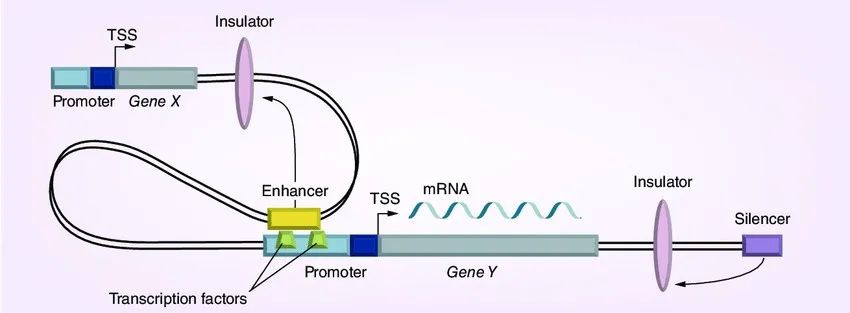
所以,做转录因子的 ChIP-seq,目的就是找到这个“开关”蛋白究竟控制了基因组上的哪些基因。
方向二:研究组蛋白修饰 (Histone Modification)
在我们的细胞核里,长长的 DNA 是缠绕在组蛋白上的。组蛋白可以被加上各种化学“标签”(如甲基化、乙酰化),这些“标签”会影响 DNA 的包裹松紧度,从而调控基因的表达。这就像给基因表达上了一个“音量调节器”。
- 组蛋白乙酰化 (ac):通常让染色质变“松”,基因更容易表达,相当于“调高音量”。
- 组蛋白甲基化 (me):情况比较复杂,有的类型是激活转录(如H3K4me3),有的则是抑制转录(如H3K27me3、H3K9me3),相当于“调高或调低音量”。
这里的 H3K4me3 表示在第3号组蛋白(H3)的第4个赖氨酸(K)上有3个甲基化(me3)修饰。
.jpg)
所以,做组蛋白修饰的 ChIP-seq,目的就是查看基因组上哪些区域的“音量”被调高了,哪些被调低了,从而了解基因的表达调控状态。
2. ChIP-seq 的实验流程是怎样的?
整个实验过程可以简化为“固定-打碎-沉淀-纯化-测序”五步曲。
.jpg)
- **交联固定 (Crosslinking)**:用甲醛处理细胞,把蛋白质和它正结合着的 DNA “粘”在一起,防止它们在后续操作中分开。
- **打碎 DNA (Fragmentation)**:用超声波或酶将长长的染色质打断成小片段。
- **免疫沉淀 (Immunoprecipitation)**:加入针对我们目标蛋白的特异性抗体(就像一个精确制导的钩子),这个抗体就会抓住目标蛋白以及与之相连的 DNA 片段。
- **纯化 DNA (Purification)**:洗掉其他杂质,并将蛋白质消化掉,只留下被目标蛋白“抓住”的那些 DNA 小片段。
- **建库测序 (Sequencing)**:将这些 DNA 片段进行二代测序,读取它们的序列信息。
3. 如何看懂 ChIP-seq 的结果图?
测序完成后,分析才是重头戏。我们通常会经历质控、序列比对、Peak calling 和功能注释这几个步骤。其中,我们在文章里最常看到的结果图,主要来自后两个步骤。
.jpg)
核心概念:Peak (峰)
我们将测序得到的无数 DNA 短片段比对回参考基因组上。如果某个蛋白在特定位置与 DNA 结合,那么就会有很多来自该位置的 DNA 片段被测序。当把这些片段“堆”在基因组坐标轴上时,就会形成一个像山峰一样的凸起,我们称之为 Peak。
Peak 越高,意味着蛋白和该区域结合的信号越强。
.jpg)
Peak 的形状有讲究:
- 转录因子 的结合位点非常精确,其 Peak 形状通常是又高又窄的“尖峰”。
- 组蛋白修饰 往往覆盖一片区域,其 Peak 形状则是起伏平缓的“宽峰”。
.jpg)
常见结果图解读
1. Peak 在基因组上的分布(Genomic Annotation)
这张图告诉我们,目标蛋白主要喜欢结合在基因组的哪些功能区域。比如,它是不是更倾向于结合在启动子区(Promoter,基因的“开关”区域)、内含子(Intron)还是外显子(Exon)?
.jpg)
2. Peak 在转录起始位点(TSS)附近的富集图
由于基因调控的关键区域通常在转录起始位点(TSS)附近,所以研究者们特别关心蛋白在 TSS 周边的结合情况。这类图通常由两部分组成:
- 峰图(左):展示了所有基因 TSS 位点上下游区域信号的平均强度,可以看出蛋白结合的总体趋势。
- 热图(右):展示了每个基因 TSS 位点周围的信号强度,颜色越深代表信号越强。
.jpg)
3. 基因功能富集分析(GO / KEGG)
通过分析 Peak 附近的基因(即蛋白可能调控的目标基因),我们可以了解这些基因主要参与哪些生物学过程(GO分析)或信号通路(KEGG分析)。这能帮助我们从宏观层面理解该蛋白的功能。
.jpg)
4. 基序分析 (Motif Analysis) 的 Logo 图
对于转录因子,它识别并结合的 DNA 序列通常有特定规律,这个规律序列就叫 Motif。通过分析所有 Peak 区域的 DNA 序列,可以找出这个共同的 Motif。
Logo 图就是 Motif 的可视化展示:
- x 轴代表序列位置。
- 每个位置上堆叠的字母代表可能的碱基(A/T/C/G)。
- 字母越大,说明转录因子在这个位置上越倾向于结合该碱基。
.jpg)
通过与已知数据库(如 JASPAR)比对,我们甚至可以推断出这个结合蛋白究竟是哪个转录因子。
5. 多个 ChIP-seq 结果的关联分析
文章中还可能出现韦恩图(Venn Diagram)等,用来比较不同蛋白(或同一蛋白在不同条件下)结合的靶基因有哪些重叠,从而探索它们之间是否存在协同或拮抗的关系。
.jpg)
参考文献