LD衰减图
LD衰减图就是利用曲线图来呈现基因组上分子标记间的平均LD系数随着标记间距离增加而降低的过程。
LD系数
- 不同基因座间的相关性,用一个数值来衡量就是D值。类似相关系数是标准化后的协方差,LD系数(r^2)则是标准化后的D值(图2中有计算公式),这个数值在0~1波动。r^2=0就是两个位点完全不相关,群体中单倍型分布是随机的(观测值=期望值)。r^2=1就是两个位点完全相关,某些基因型(A)只与特定的基因型(B)共同出现。如下图所示:

- 一般而言,两个位点在基因组上离得越近,相关性就越强,LD系数就越大。反之,LD系数越小。也就是说,随着位点间的距离不断增加,LD系数通常情况下会慢慢下降。这个规律,通常就会使用LD衰减图来呈现。下图为黄瓜群体遗传分析文章中各个亚群的LD衰减图:
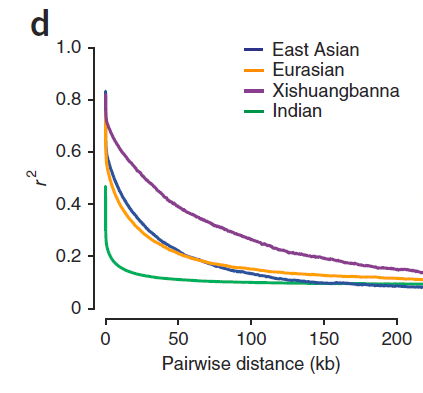
参数解释
横坐标是物理距离(kb),纵坐标是LD系数(r^2)。
LD衰减距离:当平均LD系数衰减到一定大小的时候,对应的物理距离。
定义:r²降至特定阈值(常取 0.5 或 0.2)时的物理距离
衰减距离 群体历史推断 对GWAS的影响 短 古老群体、无瓶颈 需高密度SNP芯片 长 近期瓶颈、人工选择 可用低密度SNP
衰减速率:单位距离内r²的下降值(如 Δr²/kb)
- 斜率绝对值大 → 重组活跃(如端粒区域)
- 斜率绝对值小 → 重组抑制(如着丝粒区域)
整体衰减模式:陡峭 vs 平缓
1. 曲线陡峭下降
- 特征:
- 短距离内(如 < 10 kb)r² 从接近1迅速降至接近0
- 衰减距离短(例如 r²=0.5 时距离 ≤ 20 kb)
- 生物学意义:
- LD衰减快(衰减距离短)则需要非常高的标记密度(如测序) 才能达到相同的覆盖度,但定位精度更高(定位区间较小)。
- LD衰减快则有助于将信号精确定位到更小的基因组区间。
- LD的快速衰减表明snp的亲缘关系非常接近。因此,使用的snp越丰富,GWAS的结果在某种程度上就越精确(
- 高重组率:基因组重组事件频繁,打破LD的速度快
- 大有效群体规模:群体历史悠久、近交程度低(如野生种群)
- 无近期选择压力:未经历人工选择或瓶颈效应
2. 曲线平缓下降
- 特征:
- r² 随距离下降缓慢,长距离(如 > 100 kb)仍维持较高值(r² > 0.2)
- 衰减距离长(例如 r²=0.5 时距离 ≥ 100 kb)
- 生物学意义:
- LD衰减慢(衰减距离长)意味着较稀疏的标记密度(如芯片)就能覆盖基因组,定位到目标性状位点(但定位区间较大)。
- LD衰减慢会降低定位精度,找到的显著信号区域通常较大(包含多个连锁的SNP)。
- 低重组率:染色体区域重组抑制(如着丝粒附近)
- 小有效群体规模:群体经历瓶颈、奠基者效应或人工选择
- 近期驯化/选择:农业动植物(如玉米、奶牛)因人工选择延长LD
局部曲线特征解析
| 特征区域 | 典型特征 | 形成原因 | 应用/意义 |
|---|---|---|---|
| 初始平台区 | 曲线起始段高位(r²≈1),几乎无下降 | 1. 物理距离极近(< 1 kb),重组概率趋近于 0 2. 强连锁不平衡(LD)区域的单倍型区块保守性 | GWAS 中设计 Tag-SNP(用少数 SNP 代表整个区块) |
| 异常抬升区 | 特定距离点(如 50-100 kb)出现 r² 回升凸起 | 1. 平衡选择区域(如 MHC 基因区) 2. 结构变异(倒位、重复序列抑制重组) 3. 技术假象(样本量不足或分型错误) | 提示潜在功能区域或数据质量问题 |
| 阶梯式下降区 | 曲线呈分段陡降(非平滑衰减) | 1. 重组热点(如 PRDM9 蛋白结合位点) 2. 染色体边界(常染色质与异染色质过渡区) | 反映基因组重组事件的热点分布 |
技术因素对曲线的影响
| 因素 | 对曲线的影响 | 解决方案 |
|---|---|---|
| 样本量不足 | 曲线波动大、r²高估 | N ≥ 50个体 |
| SNP密度低 | 低估远距离LD | 测序深度 > 10× |
| 群体亚结构 | 整体LD抬升(假阳性) | PCA校正群体分层 |
绘制方法
LD衰减图的绘制,实际上有两个步骤:
1)计算marker间两两的LD系数大小
这个可以使用haploview软件完成。计算的时候,只要设定一个关键的参数:区间大小。例如设定为5Mb,那么软件就会计算基因组上所有距离<5Mb的两两位点间的LD系数。实际上这个参数设定更大也没有意义,一般情况下位点间的相关性不会延伸到大于5Mb这么远的距离。
2)绘图
将LD系数按照对应的两个marker间的距离进行分类,例如:距离按照区间大小05k,5k10k,10k~15k…..分别分类。如果重测序的数据,SNP标记密度较大,这个分类区间可以设置小一些;如果是简化基因组数据,SNP标记较为稀疏,则分类区间可以适当加大。然后计算每种距离分类的LD系数的均值。最后在利用均值绘制曲线图就ok了。这一步的绘图,使用excel或R语言都可以轻松完成。
LD衰减图
https://oldstory.cn/2025/08/13/ld_shuai_jian_tu/